郑敏课题组在Nucleic Acids Research上发文报导新型gRNA文库高通量合成技术——CTDE
2023年3月20日,银河线路检测中心附属第一医院传染病诊治国家重点实验室郑敏团队与生命科学研究院张龙团队在国际核酸专业刊物《Nucleic Acids Research》上发表题为“A new method to synthesize multiple gRNA libraries and functional mapping of mammalian H3K4me3 regions“的研究论文。
哺乳动物的基因组由数亿个碱基对组成,其中非编码区域约占据99%。在CRISPR-Cas9技术问世之前,对这些区域的功能进行注释一直是生命科学领域的难题。随着CRISPR-Cas9技术的出现,基因组序列编辑变得更加精准和便捷。然而,由于整个基因组序列的庞大,使用饱和突变筛选方法对非编码基因组进行注释时需要合成大量的文库。因此,开发高效低成本的gRNA文库合成方法对促进基因组注释的发展至关重要。本团队创新建立了高保真、高覆盖度、高效率、低成本新型gRNA文库高通量合成技术——CTDE(Controlled Template Dependent Elongation)方法,该方法是一套依赖于可逆终止子系统的合成技术,该技术可利用任意DNA作为模板,合成出整个gRNA文库。CTDE合成出的文库长度统一,且忠实于模板DNA,这为非模式生物或有突变的基因组进行筛选功能注释铺平了道路。
该研究首次实现了鼠胚胎干细胞和人肝癌细胞H3K4me3区域的生存必需元件的注释,并解析了这些元件上游的调控转录因子。作为示例,该研究发现长非编码RNA LINC00339受到其内含子的调控,并揭示该LncRNA是一个新的肝癌细胞周期调控因子,影响肝癌细胞的增殖,可作为肝癌诊断和治疗的新靶点。
CTDE技术高效且成本低,可对感兴趣的核酸进行文库的构建,为解析哺乳动物全基因组序列功能的探究提供了一种新的解决方案。本文的第一作者及共同第一作者分别是博士研究生潘辰、李然、水丽燕和肖政云,郑敏、贾俊岭和张龙教授为通讯作者。本研究获得国家自然科学基金、浙江省重点研发项目的经费支持。
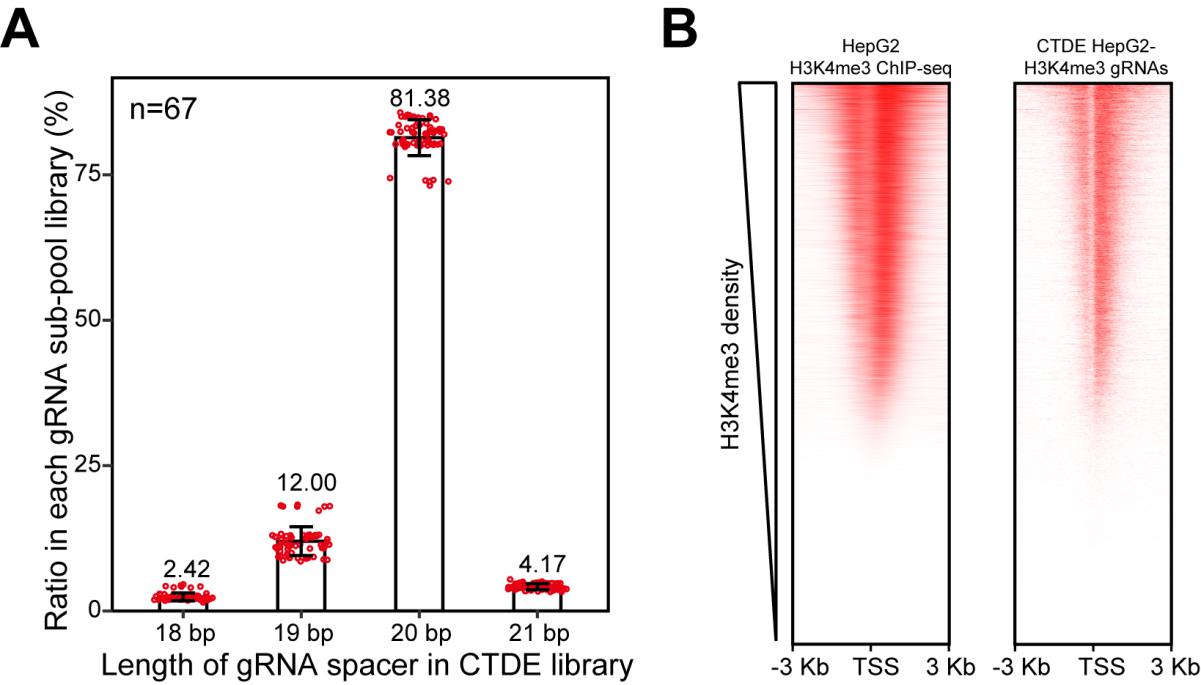
图示1:A. 67个HepG2细胞H3K4me3区域文库中gRNA的长度统计分析,这些文库采用CTDE法进行构建;B. CTDE-HepG2-H3K4me3 gRNA文库与模板DNA在基因组中分布的热图。
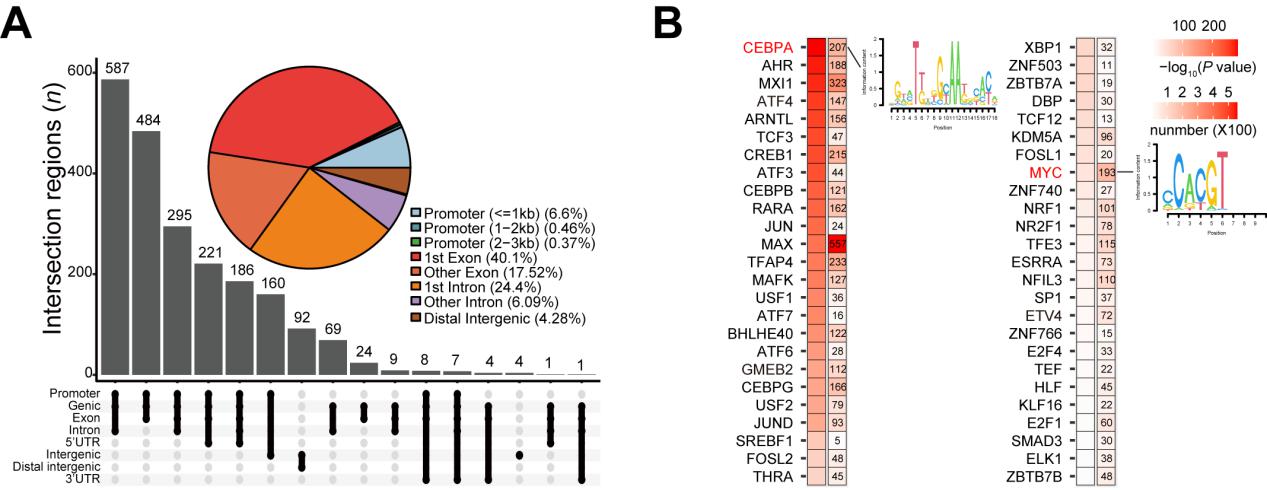
图示2:A. HepG2细胞必要元件在基因组中的分布分析结果;B. HepG2必要元件上游转录因子分析。
原文链接:https://academic.oup.com/nar/advance-article/doi/10.1093/nar/gkad198/7080829